Macrostomum lignano因为其身体具有极强的自愈能力是研究干细胞再生及分化的重要模式物种()。
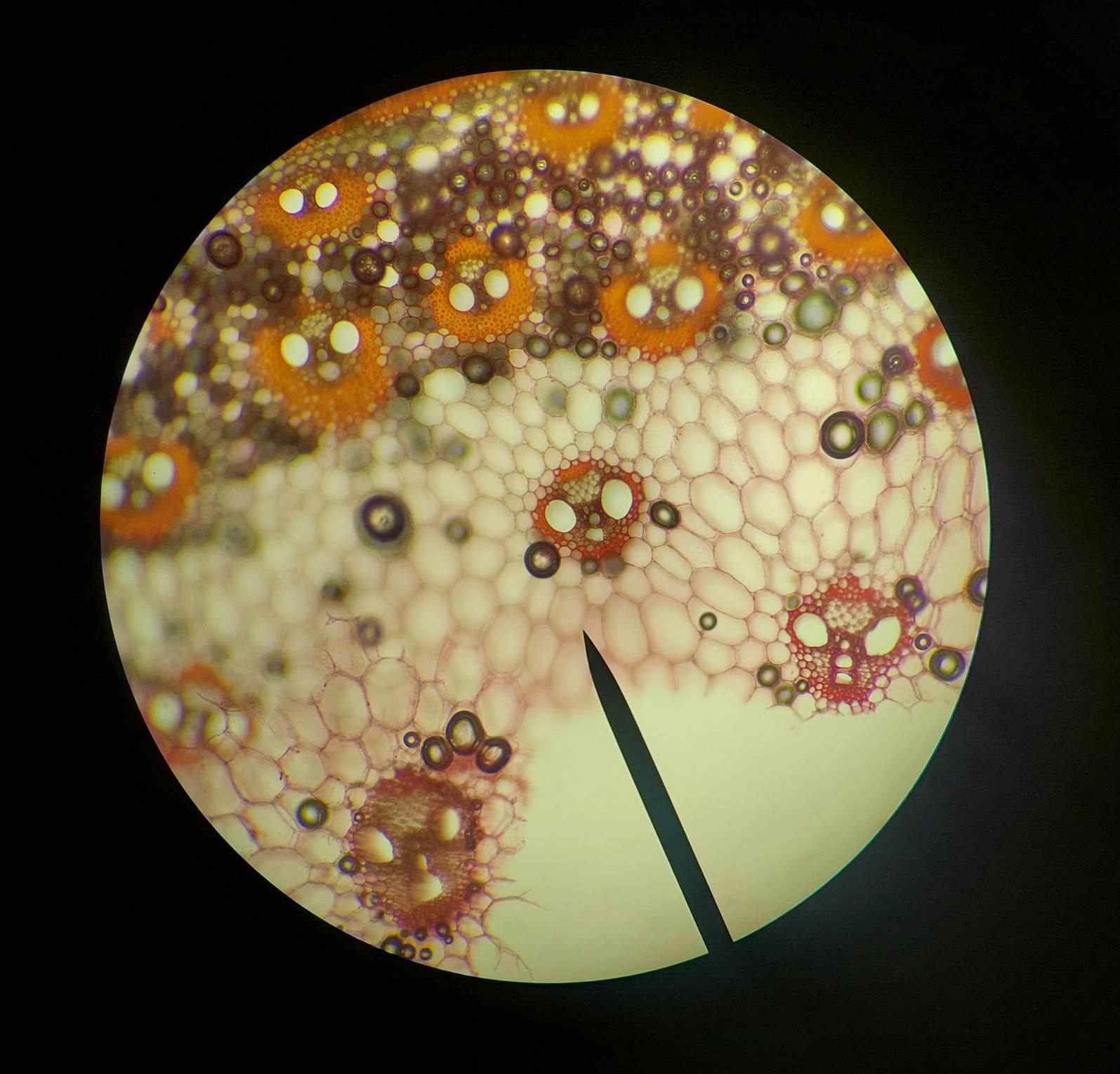
本研究中对一个M. lignano近亲繁殖 35代的DV1系首先进行了170X的二代测序,由于~75%的基因组由简单重复序列和转座子组成,组装后的基因组很碎,contig N50 只有222bp,最长的contig只有144kb。为了改善组装的结果,研究者增加了21X的PacBio数据,最终contig N50的长度达到了64Kb,最长的contig由114Kb延伸到了627Kb,contig N50的长度达到64Kb。
将ESTs和BACs reads 比对与基因组比对,发现92%的ESTs和91%的BACs reads都以超过90%的相似度比对到基因组上;取部分只由二代数据组装的contig与只由三代组装的基因组比对,发现相似度超过99%,说明M. lignano组装结果的可靠性。
分析发现, 55%的contig的末端50%的碱基都是富含GA的简单重复,20-24碱基的重复占主导,串联重复的长度超过100bp,这些串联重复遍布整个基因组,因此尽管加入21X的Pacbio数据对基因组组装的改善程度仍然有限。
串联重复被认为与CpG甲基化有关,但该研究中在M. lignano只检测到了低水平的甲基化。分析基因组的重复序列类型,发现目前已知的转座元件只占基因组的0.17%,而23,064种新型的重复序列占到了基因组的~51%,这些重复序列的平均长度为946bp,最长的达到了20Kb,这些新型的重复序列只有1,693个得到了注释,因此M. lignano基因组中可能存在大量的新型的转座子。研究者用248个保守真核基因与组装后的基因组比较,结果显示M. lignano基因区的组装结果较为完整,有19,794个基因获得了转录组数据支持。
M. lignano分类地位及身体结构
转录组测序数据组装得到149,647个转录本,99.47%的转录本可以比对到基因组,64,842个转录本(占总数的43.3%)获得注释,这些注释的转录本中有5%注释到转座子暗示基因组内存在活跃的转座子。
研究者发现有7,500个转录本在5’端存在splice leader (SL),最长的SL达到45nt,长转录本似乎容易拥有长的SL,这些长SL其它涡虫有高的相似性但长度是其它涡虫的两倍以上。有意思的是M. lignano与人共有约2000个基因,但线虫和果蝇中却丢失了这些基因。
先前的一些研究证明转录因子如Oct4/Pou5f1, Nanog, Klf4, c-Myc, and Sox2在维系干细胞全能性方面起重要的作用,但M. lignano基因组只发现了Sox2,尽管这样,与维系干细胞全能性的路径如Jak-Stat,Wnt,MAPK等却具有很好的保守性。
在M. lignano基因组中发现了来自11个大类的49个同源异形盒基因,这些同源异形盒基因有些发生了物种特异的扩张如:Hox1,HK2.2,Irx等,同时研究者还发现了一些在其它已测序扁形动物中没有发现的同源异形盒家族,如Cdx,Dbx,Prrx等与再生有很大联系。
为了研究基因表达与再生之间的关系,研究针对扁虫的不同部位进行了切割,观察不同时间段下基因的差异表达()。
结果显示3-12个小时内应答上调都是一些生长因子,这些生长因子参与细胞的生长和分化,一些参与细胞信号的激酶、ATP酶、GTP酶基因也发生了上调。随后参与细胞组织如细胞黏附、外伤应答、细胞骨架的相关在后期发生上调。在24-48小时,含有锌指结构域的因子、Klf因子TNF类蛋白基因参与了上调。
最后研究者通过分析48小时后下调的基因发现了一个可能参与再生的决定因子Smad4。
M. lignano再生发生时不同时间内基因差异表达热图
组装方案推荐
纯三代20K文库测序80X + 二代小文库测序50X + 光学图谱测序150X-200X
二代小文库50X测序数据作用:用于调研图与后期三代基因组纠错
纯三代20K文库80X测序数据作用:用于兆级contig组装
光学图谱150-200X测序数据作用:用于组装纠错与super-scafflod组装
友情推荐
现在同时具有三代测序平台与光学图谱测序平台,并且在复杂基因组组装与文章运作方面势力较强的高通量测序公司为大家推荐两个:
1)北京百迈客:RSII平台 + BioNano平台(芥菜文章章)
2)深圳华大基因:RSII平台 + BioNano平台
听说上述两家公司都在引进新一代三代测序平台squel,这将进一步提升三代测序的通量,不过听说现在squel平台的读长与质量还存在问题,预计在2016年都无法解决,大家在选取测序平台时尽量先选取RSII平台,等squel平台稳定后再进入。
Genome and transcriptome of the regeneration-competent flatworm, Macrostomum lignano
Abstract
The free-living flatworm, Macrostomum lignano has an impressive regenerative capacity. Following injury, it can regenerate almost an entirely new organism because of the presence of an abundant somatic stem cell population, the neoblasts. This set of unique properties makes many flatworms attractive organisms for studying the evolution of pathways involved in tissue self-renewal, cell-fate specification, and regeneration. The use of these organisms as models, however, is hampered by the lack of a well-assembled and annotated genome sequences, fundamental to modern genetic and molecular studies. Here we report the genomic sequence of M. lignano and an accompanying characterization of its transcriptome. The genome structure of M. lignano is remarkably complex, with ∼75% of its sequence being comprised of simple repeats and transposon sequences. This has made high-quality assembly from Illumina reads alone impossible (N50 = 222 bp). We therefore generated 130× coverage by long sequencing reads from the Pacific Biosciences platform to create a substantially improved assembly with an N50 of 64 Kbp. We complemented the reference genome with an assembled and annotated transcriptome, and used both of these datasets in combination to probe gene-expression patterns during regeneration, examining pathways important to stem cell function.
原文:基因组
欢迎关注